

当前位置:首页 > RNA测序服务
Small RNA是生物体内一类序列长度在18~30bp,对生命活动具有重要调控作用的小分子RNA,主要包括miRNA、piRNA、siRNA。他们通过与靶mRNA特异性结合来调控靶mRNA的表达,从而在疾病发生和生长发育过程中起着重要的调控作用。小RNA测序是利用高通量测序技术对小RNA进行检测,能快速鉴定出不同组织、不同发育阶段、不同疾病状态下已知和未知的小RNA 的表达水平及其表达差异,为研究小RNA的功能及调控机制提供有力的工具。

|
样本要求 |
测序策略 |
交付周期 |
|
样品类型: Total RNA 样品浓度: ≥200 ng/μl 样品总量: ≥5μg 样品纯度:OD260/280为1.8~2.2,OD260/230为 1.8~2.2;28S:18S≥1.5;RIN≥8 |
测序模式:Hiseq3000/4000 (SE 50) 测序深度:10M/20M clean reads
|
45天 |
(1)使用小RNA测序鉴定猪完整发育阶段的microRNAome
研究背景:
猪由于其个体大小,解剖学,生理学,新陈代谢,病理学以及药理学上与人十分相近,已成为一种重要的研究人类疾病的模式动物,将来有可能成为人类异种移植的重要器官来源。
研究内容:
研究人员收集了猪从胚胎到成年共计10个发育阶段的样品,覆盖了猪发育的全部代表性时期,系统深入地发掘了猪microRNAome图谱。
研究结果:
研究人员对测序得到的猪microRNAome进行了详尽的生物信息学分析,给出了有关miRNA的末端序列变异、miRNA前体结构、染色体定位、特定发育阶段的miRNA表达以及miRNA保守性等详细信息。

图1. 猪保守miRNA(灰色)和特异miRNA(黑色)在每个发育时期的数量比例
(2)利用small RNA 测序技术分析鉴定小立碗藓的siRNA形成机制
研究背景:
多数植物的small RNA是在序列上特异性靶向mRNA的负调控子。在被子植物中,具有两种含量非常丰富的內源性small RNA分子,分别是长度约21 nt的miRNAs和24 nt的siRNAs,其中siRNAs来源于重复序列,能增强靶向位点的DNA甲基化程度。但在其他陆地植物中,siRNAs特性还不明晰。
研究内容:
为了分析小立碗藓的miRNAs和siRNAs特性,研究人员利用small RNA 测序结合WGBS技术检测小立碗藓的野生型和突变体,鉴定小立碗藓的siRNAs和miRNAs特征表达谱,并分析了siRNAs形成位点与甲基化 的关系,结果表明:mDCL参与了小立碗藓內源siRNAs形成,促进23 nt的siRNAs富集;虽然siRNAs对其形成区域DNA甲基化 的改变很小,但抑制了体内LTR逆转录相关基因的表达。
研究结果:
分析鉴定1090个可以形成长度23-24 nt siRNAs的位点,这些位点绝大多数在DNA甲基化 富集的基因间区。
新的miRNA 注释方法鉴定获得130个miRNAs,其中112个是已知的、3个是已知miRNA家族的新成员、15个来源于新的
miRNA家族;比较新老注释方法,发现两种注释方法共同分析获得的有114个miRNA s。
对小立碗藓的mdcl突变体分析发现,mDCL参与了小立碗藓的內源siRNA形成,促进23 nt的siRNAs富集。
经过WGBS分析显示,尽管小立碗藓的內源siRNA对形成区域的DNA甲基化 改变很小,但抑制了体内LTR逆转录相关基因的表达。
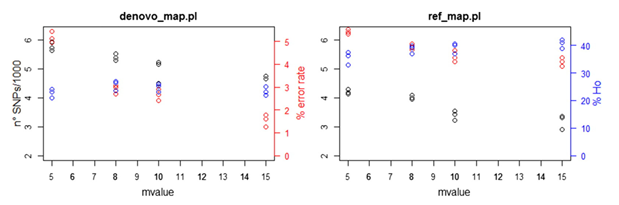
图1. 比较在denovo_map.pl 和 ref_map.pl SNPs的差异
下一篇:转录组测序
上一篇:长链非编码RNA测序