RNA免疫共沉淀测序
发布时间 2017-09-14
RNA Immunoprecipitation (RIP) 是研究细胞内RNA与蛋白结合情况的技术,是了解转录后调控网络动态过程的有力工具,能帮助我们发现miRNA的调节靶点。这种技术运用针对目标蛋白的抗体把相应的RNA-蛋白复合物沉淀下来,然后经过分离纯化就可以对结合在复合物上的RNA进行测序分析。将RIP技术结合新一代测序技术,应用于癌症以及其它疾病整体水平的RNA变化检测。
|
样本要求
|
测序策略
|
交付周期
|
|
样品类型: RNA样品
样品浓度: ≥2ng/μl
样品总量: ≥50ng
|
测序模式:Hiseq3000/4000 (PE150/SE50)
测序深度:10/20M clean reads
|
60天
|
与RIP芯片相比,可在全转录组范围对蛋白结合位点进行筛选与鉴定,且敏感度高;
可获得高水平的信噪比数据,准确区分真实事件与噪音,精确定位蛋白结合位点;
深度测序保证了检测随机性,可不需技术重复。
RIP-seq研究RNA甲基化调控RNA二级结构影响RNA与蛋白质相互作用
研究背景:
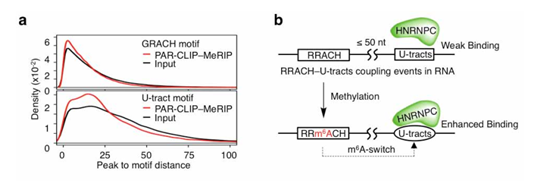
RNA分子腺嘌呤第6位氮原子上的甲基化修饰(N6-methyladenosine,m6A)是高等生物RNA上含量最为丰富的一种RNA表观修饰形式,这种表观修饰会参与到很多转录后调控过程中,与多种生理功能有关,是一类极为重要的表观修饰。一个与肺癌转移相关的lncRNA(MALAT1)的茎环结构上具有一个m6A修饰位点2577-A/m6A,该位点修饰状态会改变数种核酸酶对MALAT1的消化能力,从而影响RNA的稳定性。HNRNPC蛋白能特异结合新生mRNA多聚尿嘧啶区域影响其靶点的稳定性、剪切、运输及翻译过程。
研究内容:
1. CLIP-MeRIP分析
针对HEK293T,METTL3KD两个细胞系进行HNRNPC特异抗体CLIP,分离RNA进行MeRIP,构建small RNA文库,SE50测序。分析甲基化转移酶(METTL3)缺失抑制RNA发生m6A甲基化对蛋白质HNRNPC结合RNA能力的影响以及结合位点的序列偏好性,研究结果证实,RNA甲基化能够增强HNRNPC结合RNA的能力(图1)。
2. RNA甲基化调控基因分析
针对HEK293T,HNRNPC KD , METTL3 KD, METTL14 KD四个细胞系进行RNA-seq,筛选RNA甲基化调控基因。有接近1000个包含m6A开关的转录本出现变化,对这些基因进行功能富集分析,GO分析表明这些转录本所对应的基因与细胞增殖等生物过程有关,说明RNA甲基化能够影响肿瘤的增殖,并通过体外试验验证该结论。
研究结论:
转录后的m6A修饰可以通过调控RNA-HNRNPC互作来影响编码以及非编码RNA的结构,从而进一步影响核内基因转录及成熟过程,从而影响细胞的增殖,在肿瘤的发生发展中起重要的调控作用。
1. Liu N, Dai Q, Zheng G, et al. N6-methyladenosine-dependent RNA structural switches regulate RNA-protein interactions [J]. Nature, 2015, 518(7540):560.