

当前位置:首页 > DNA测序服务
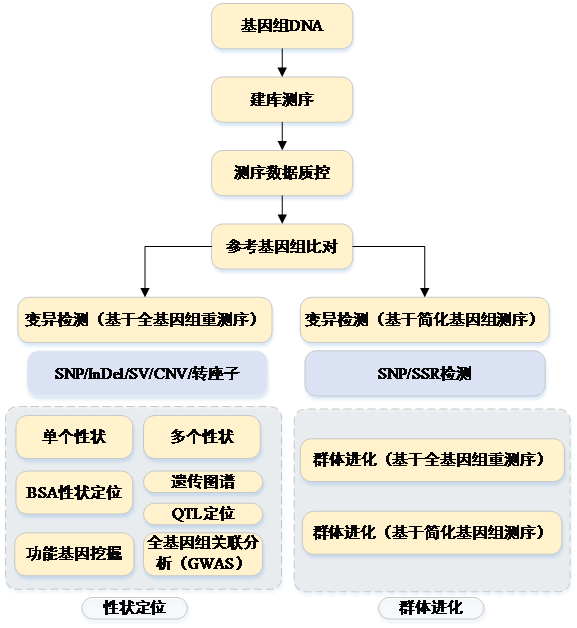
全基因组测序(Whole Genome Sequencing,WGS),是对全基因组进行测序扫描,能在全基因组范围内解读所有常见和稀有变异信息,进行单核苷酸多态位点(Single Nucleotide Polymorphisms, SNP)、插入缺失位点(Insertion Deletion, InDel)、结构变异(Structure Variation,SV)以及拷贝数变异(Copy Number Variation, CNV)深度挖掘,进而全面揭示基因组突变类型与染色体重排事件,对疾病机理研究、药物靶点筛选等具有指导意义。应用范围涉及临床医药研究、群体遗传学研究、关联分析、进化分析等众多领域。
|
样本要求 |
测序策略 |
交付周期 |
|
样品类型: 基因组DNA 样品浓度: ≥20 ng/μl(Qubit定量) 样品总量: ≥3.0 μg(3次建库总量,不包括样品检测损耗,Qubit定量) 样品纯度: OD260/280为1.7~2.2 电泳要求: 主带清晰,无降解或轻度降解,无严重RNA、蛋白质污染(距送样日最近的电泳结果)。 |
测序模式:Hiseq3000/4000 测序深度:推荐30X
|
60天 |
1. Huang X, Kurata N, Wei X, et al. A map of rice genome variation reveals the origin of cultivated rice[J]. Nature, 2012, 490(7421):497-501.