热搜词:宏基因组测序 芯片检测

发布时间:2018-05-20 17:00:00 点击数:次
核糖体印记测序Ribosome Profiling (即Ribo-seq) 是针对RNA到蛋白质的翻译过程的研究。通过高通量测序技术研究被核糖体保护的30nt左右的mRNA片段,对此片段进行深度测序分析。从转录后水平分析翻译效率,揭示转录组与蛋白组结果不一致的真相。
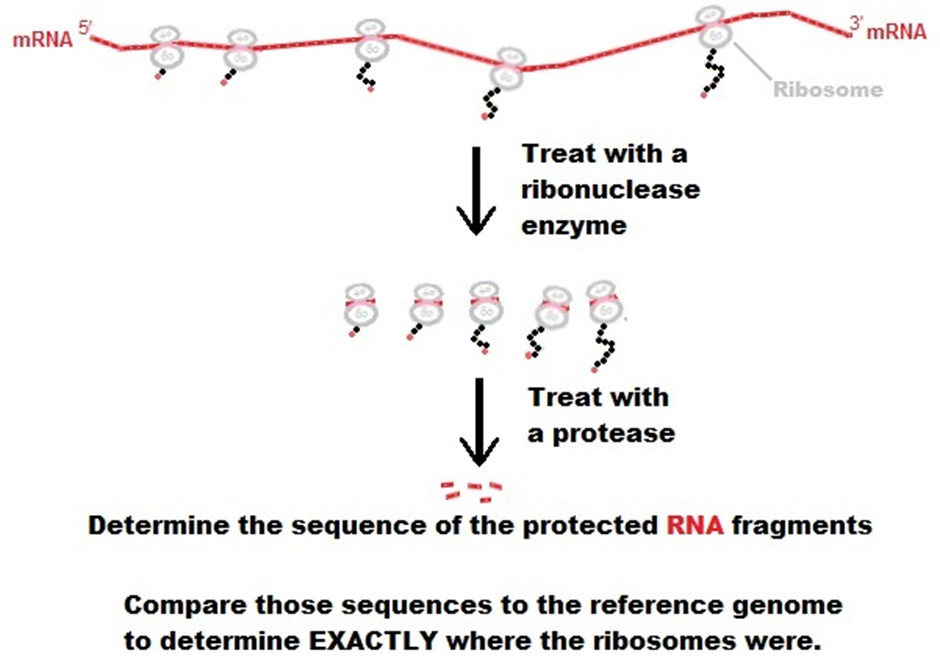
1、提取total RNA
2、用核糖核酸酶进行消化
3、消化蛋白
4、获取被核糖体保护的RNA
5、建库测序
目前翻译组研究中主要存在两种技术,分别为 RNC-seq与 Ribo-seq。二者之间区别主要在于 RNC-seq 目标是全长翻译中的mRNA 测序,而 Ribo-seq 是把 mRNA 中被核糖体包裹的所有片段获取下来,非整段 mRNA。因此,Ribo-seq 更能准确预测蛋白翻译的丰度,技术上更为精准。
Ribosome Nascent-chain Complex(RNC)翻译组测序RNC = Ribosome Nascent-chain Complex, 研究翻译的总体叫做翻译组学。翻译组包括广义的翻译组和狭义的翻译组,广义的翻译组指的是直接参与翻译过程的所有元件,包括核糖体、正在翻译的mRNA、tRNA、调控性RNA (如miRNA, lncRNA等)、新生肽链(nascentpolypeptide chain )、各种翻译因子等(图2);狭义的翻译组特指正在翻译的mRNA。传统的转录组测序,是对细胞中所有的mRNA进行测序,无论有没有在翻译;而翻译组测序则是对直接参与翻译过程的所有元件进行测序,比如对卡在核糖体上正在翻译的mRNA(RNC-mRNA)进行测序等(图2)。

翻译组与转录组测序的区别
1. 探究翻译机制:
(1) 关注正在翻译的基因中,哪些处于高效翻译,调控翻译效率的元件有哪些?
(2) 解释双解码的发生机制,哪些基因可以发生双解码?具有哪些结构特征?
(3) 探究非常规翻译现象。
2. 深化转录组测序分析:
(1) 关注差异表达的基因中,究竟有哪些正在翻译,且翻译效率如何?
(2) 差异表达基因太多,则缩小关注的范围(只关注正在翻译的基因)。
(3) 差异表达基因太少,则通过Ribo-seq发现更多差异表达/翻译基因。
3. 解释转录组与蛋白组结果的不一致:
(1) 转录组显示有差异或差异显著,蛋白组无差异或差异不显著,可能因为基因转录而不翻译或者翻译效率低。
(2) 蛋白组仅能粗略定位到基因区域,但是无法明确转录本,更无法明确转录本中的编码性ORF。
4. 深化非编码RNA测序分析:
(1)包括 lncRNA中的TUCP究竟是否具备编码能力。
(2)以及 circRNA对应线性RNA是否具备翻译功能/circRNA开环是否在翻译。
局限性:
当前,翻译组学的两大主要研究手段是为正在翻译中的mRNA测序(RNC-seq)和核糖体印记测序(Ribo-seq),然而这两种实验方法都极其困难,成本也十分高昂,鲜有完整的翻译组学数据可供科研人员参考。
虽然此前也存在少数Ribo-seq数据库,但其并不包含RNC-seq数据,也不包含所对应的mRNA-seq数据,因此十分局限。例如,Ribo-seq并不适合指导蛋白质组的研究。
此外,目前已存在的各种组学数据库,基本不具备数据分析功能,而一般的生物学或医学工作者根本没有能力去分析海量的测序数据。好不容易找到了几个不同研究中的数据,却发现他们的分析流程完全不同,数据没法整合分析。
Translatome DB是目前最全的翻译组学数据库,它共包含13个物种的2453个核糖体印记测序,10个RNC-mRNA测序, 1394个mRNA测序数据集,共计3857个数据集,几乎将NCBI中所有的翻译组相关数据集一网打尽,不管是从数据集的数量还是涵盖的物种类型来看,均可轻松秒杀其他同类型数据库!
Translatome DB:a comprehensive database and cloud-based analysisplatform for translatome sequencing data.