热搜词:宏基因组测序 芯片检测

武汉博越致和生物科技有限公司
电话:027-87705460
传真:027-87705460
地址:武汉市高新大道666号光谷生物城C6栋3楼
发布时间:2018-10-17 点击数:次
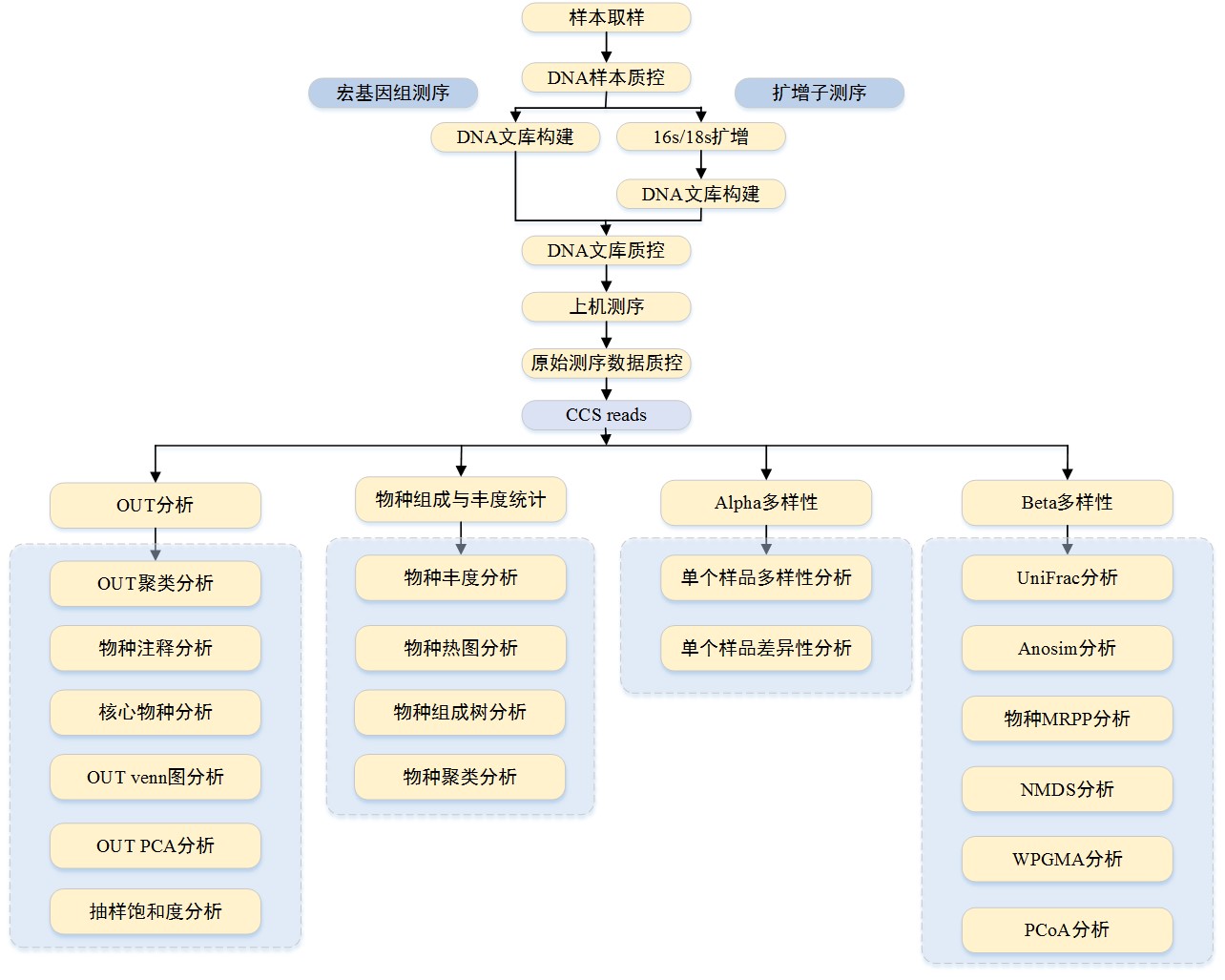
宏基因组测序是指对微生物群体进行高通量测序,分析特定环境中微生物群体基因组成及功能、微生物群体的多样性与丰度,进而分析微生物与环境、微生物与宿主之间的关系,发现具有特定功能的基因。宏基因组测序无需分离纯培养的微生物。较大扩展了微生物资源的利用,为环境微生物群落的研究提供了有效工具。宏基因组深度测序可以揭示或估计环境中真实的物种多样性和遗传多样性,挖掘具有应用价值的基因资源,应用于开发新的微生物活性物质。
而宏基因组学就是一种以环境样品中的微生物群体基因组为研究对象,以功能基因筛选和/或测序分析为研究手段,以微生物多样性、种群结构、进化关系、功能活性、相互协作关系及与环境之间的关系为研究目的的新的微生物研究方法。
宏基因组研究分两个方向:16s/18s扩增子测序和全基因组测序。
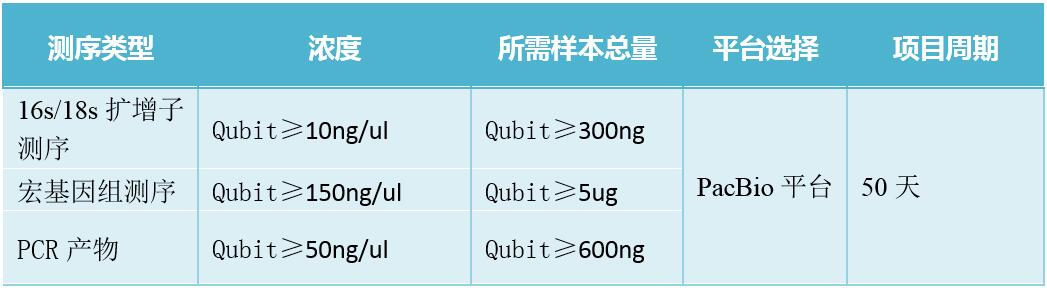
建库、测序过程无需扩增,碱基偏好性低;
数据质量高,利用PacBio CCS自我校正模式可获得高质量的数据;
长读长测序技术可以跨越全长16s区域,可有效提高环境微生物的分辨率,将更多微生物注释到种水平,提高物种丰度预测的准确性。
研究背景:
珊瑚礁是一个复杂的生态系统,由珊瑚动物和大量的相关共生体组成,包括甲藻共生菌、真菌、病毒和细菌。已有研究强调了珊瑚相关细菌的重要性及其在珊瑚存活中的基本作用。
橙黄滨珊瑚是印度西太平洋的主要珊瑚礁建造者之一。目前,关于橙黄滨珊瑚细菌群落的组成和结构知之甚少。本研究的目的有两方面:展示利用PacBio环形一致序列测定技术在微生物群落研究中的优势,并探讨印度太平洋橙黄滨珊瑚相关微生物群体的多样性和结构。这是第一次利用PacBio测序系统捕获全长16s rRNA序列进行海洋环境的宏基因组学研究。发现不同海洋区域的石珊瑚上细菌群落组成存在明显的差异性,同时也反映了基于PacBio测序的全长16s rRNA序列分析在珊瑚附着细菌群落中种的分类鉴定中的优势。
研究方法:
(1)、样本采集:采样区域位于印度西太平洋,分别采集泰国海湾(MN, TL, TA),安达曼海 (MT, SM, TC)共6个位点,每个位点取三个平行样,共18个样本;
(2)、全长16s rRNA扩增子测序:采用两步法进行全长16s rRNA扩增,第一步用16s rRNA通用引物扩增,第二步加barcode序列(图1);构建两个文库,每个文库包含9个样本,分别测12个Cells,共24个Cells,上机PacBio RSII,P6-C4;
(3)、进行全长16s rRNA数据分析。
研究结果:
(1)、24个Cells共产出28.15Gb,获得1,187,995条reads。聚合酶的平均读长为25.4Kb,CCS序列平均覆盖圈数为21.2,数据质控后共生成571,710条CCS,平均每个样本的全长16s rRNA序列的数目从20,453到60,143,平均每个样本约有31,761条(表1),质量值在99%以上;
(2)、对各样本进行OTU分析,发现所有样本中变形菌门为最优势菌群,其中Alpha、Beta、Delta、Epsilon和Gamma变形菌最为常见。但在不同海域中,丰度差异很大(图2);
(3)、通过单独进行V3-V4区域、V5-V6区域以及全长16s rRNA序列对比分析发现,99.7%的全长16s序列可以被鉴定到“种”的级别,V3-V4则只有32%-93%不等,V5-V6则只有71%-90%不等(图3);
(4)、覆盖了全长16s rRNA的序列相较于短读长获得的16s的部分可变区域而言,PacBio的全长16s分析可以更精确分析和鉴定菌群(图4)。
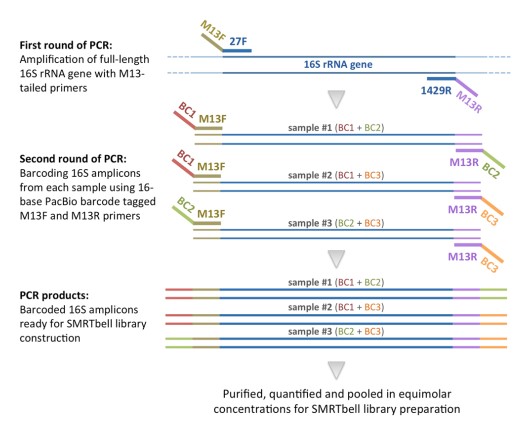
图1.全长16s扩增子扩增方法
表1.泰国海湾和安达曼海滨珊瑚中相关微生物测序的reads数统计,OTU和α多样性评估
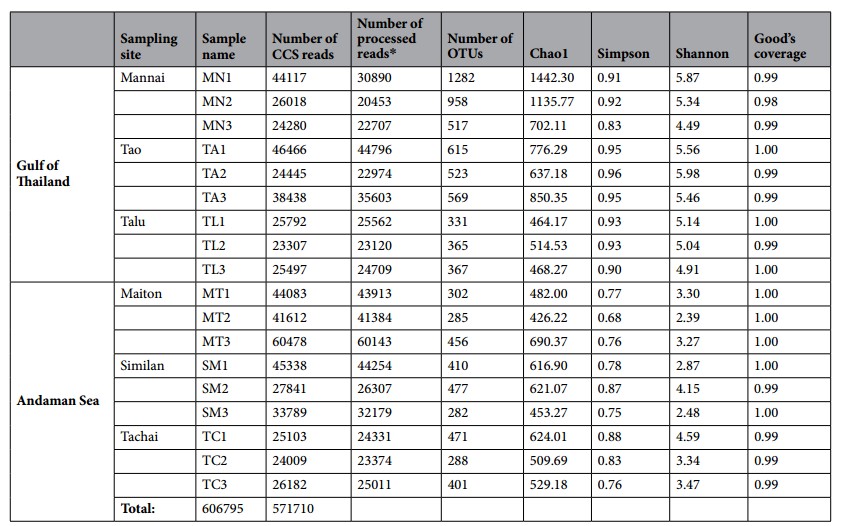
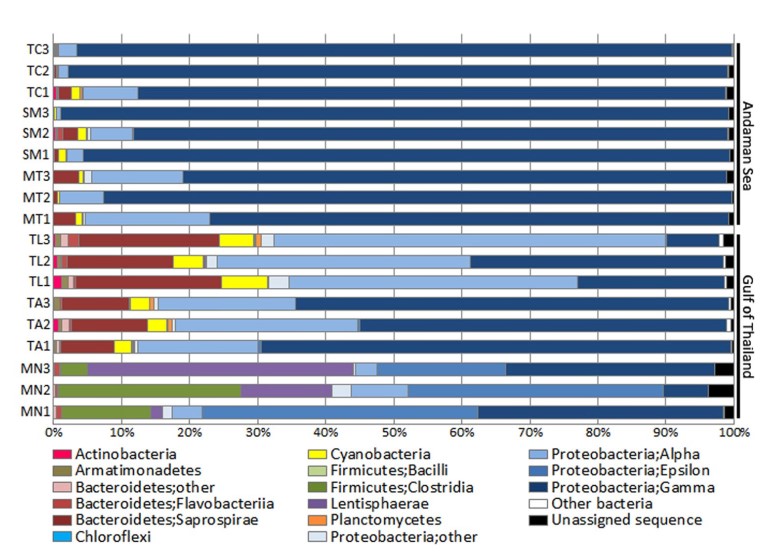
图2.用QIIME软件基于Greengenes数据库的OTUs系统分类研究
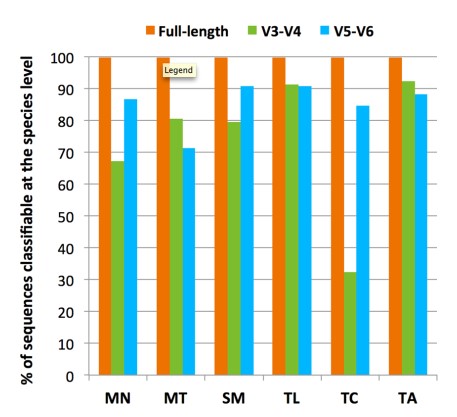
图3. 16s rRNA 全长,V3-V4、V5-V6扩增子在种分类鉴定中占比
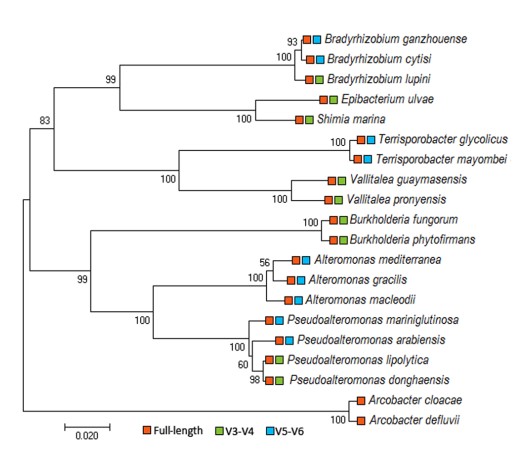
图4. 16s rRNA 全长,V3-V4、V5-V6扩增子在种分类鉴定中对比
Pootakham W, Mhuantong W, Yoocha T, et al. High resolution profiling of coral-associated bacterial communities using full-length 16s rRNA sequence data from PacBio SMRT sequencing system[J]. Scientific Reports, 2017, 7(1):2774.
