热搜词:宏基因组测序 芯片检测

发布时间:2021-08-09 10:32:58 点击数:次
基本信息

文章题目:SMOOTH-seq: single-cell genome sequencing of human cells on a third-generation sequencing platform
期刊:Genome Biology
影响因子:13.583
背景信息
单细胞全基因组测序(scWGS)是一种揭示生物样品中细胞间异质性和识别基因组变化(如拷贝数变异(CNV)和点突变)的强大工具。该技术可用于探索细胞谱系,尤其是肿瘤发生过程中细胞的演变,并能精确挖掘出批量测序中丢失的异质性信息。常见的单细胞基因组扩增方法有:DOP-PCR、多重置换扩增(MDA)技术、多次退火环状循环扩增(MALBAC)技术、和通过转座子插入的线性放大(LIANTI)技术。
目前的scWGS方法都是基于第二代测序平台,生成高度准确但相对较短的读长(数百个碱基对),非常适合于检测拷贝数变异(CNV)、小插入缺失和单核苷酸变异(SNV),但不是研究结构变异(SV)的最佳选择。SV包括缺失、插入、重复和易位。尽管它是人类体细胞遗传变异的主要来源之一,并且可能是肿瘤发生和转移的驱动力之一,但在单个细胞中很少有研究。另外,最近在人类细胞中发现了染色体外环状DNA(ecDNA),推测它在癌症发展中起重要作用,单也没有有效的scWGS方法来系统地识别它们。
近年来,单分子实时(SMRT)DNA测序技术已被用于研究人类基因组中的全基因组范围的SV。PacBio平台的HiFi模式生成的CCS读长,实现了高精度(99.8%)的长读长测序。这使得研究SV更容易和可靠。然而,SMRT DNA测序通常需要微克量的DNA来建库,而一个人类细胞只有几皮克的基因组DNA。
研究目的
目前没有有效的方法来检测单细胞全基因组水平上的结构变异(SV)和染色体外环状DNA(ecDNA)。该研究基于SMRT测序技术开发了用于单细胞基因组分析的SMOOTH-seq,利用长且高保真读长的优势实现了准确的SV检测。
研究结果
图1a为SMOOTH-seq的流程示意图。在细胞裂解和蛋白酶消化后,单个细胞的基因组DNA被低浓度的Tn5转座酶随机打断并插入接头。作者对Tn5转座酶的使用进行了优化:替换了商业化试剂中转座反应的缓冲液和PCR扩增的DNA聚合酶。反应只使用了一个Tn5接头序列来避免原始DNA片段丢失。同时,低浓度Tn5转座酶产生的长片段减少了自环化的机会。转座反应后产生的片段使用16bp标签引物进行链置换和扩增。接下来,将带有不同标签的单细胞扩增后gDNA混合在一起并纯化以制备测序文库。使用HiFi模式在Pacbio Sequel II系统上对文库进行测序,并收集CCS读长进行分析。CCS的长度超过1kb,最长可达43693bp,大部分读数约为6kb。
1. 用SMOOTH-seq检测单个K562细胞中的CNV
在分辨率为1Mb的CNV分析中,SMOOTH-seq能检测到两个K562细胞克隆之间的不同拷贝数变异(图1b)。黄色阴影突出了这两个克隆间7号和9号染色体长臂上CNV的差异。
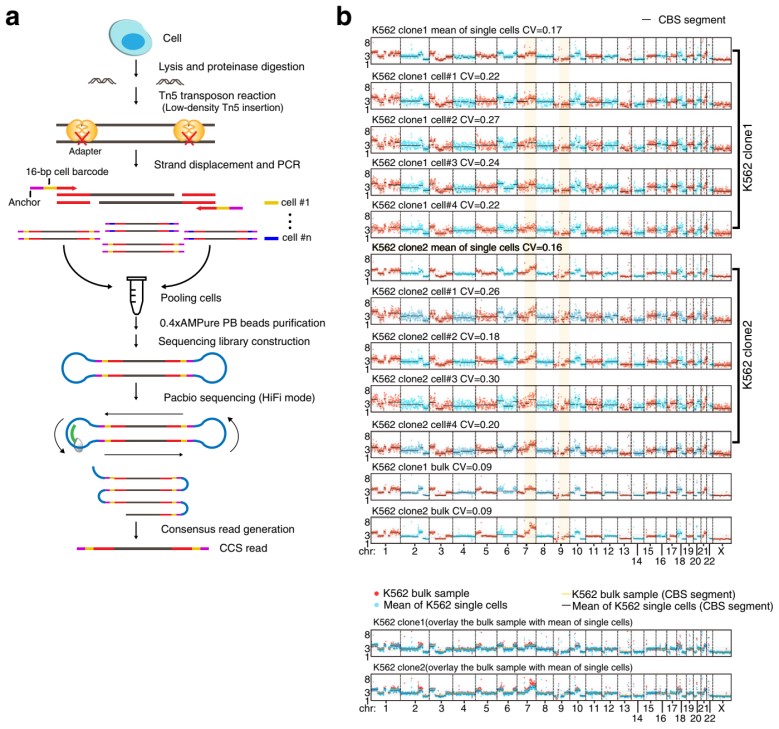
图1
2. 使用SMOOTH-seq检测SV
基因组中的SV通常可以分为缺失、插入、易位、重复和倒位(图2a)。使用K562大量细胞的基因组三代测序结果作为金标准,在两个细胞克隆中,每个单细胞的SV检测精度都很高(76.9%,平均75.2%)(图2b)。对于所有类型的SV,插入是在单个细胞中检测到的最准确的突变(87%,平均84%),缺失位居第二。重复准确率最低(不超过60%)(图2b)。随着两个克隆中支持SV事件的细胞数量增加,SV(主要是缺失和插入)的精度增加(图2c)。在不考虑测序成本的情况下,进一步提高单个细胞的测序深度将有助于提高检测SV的准确性。
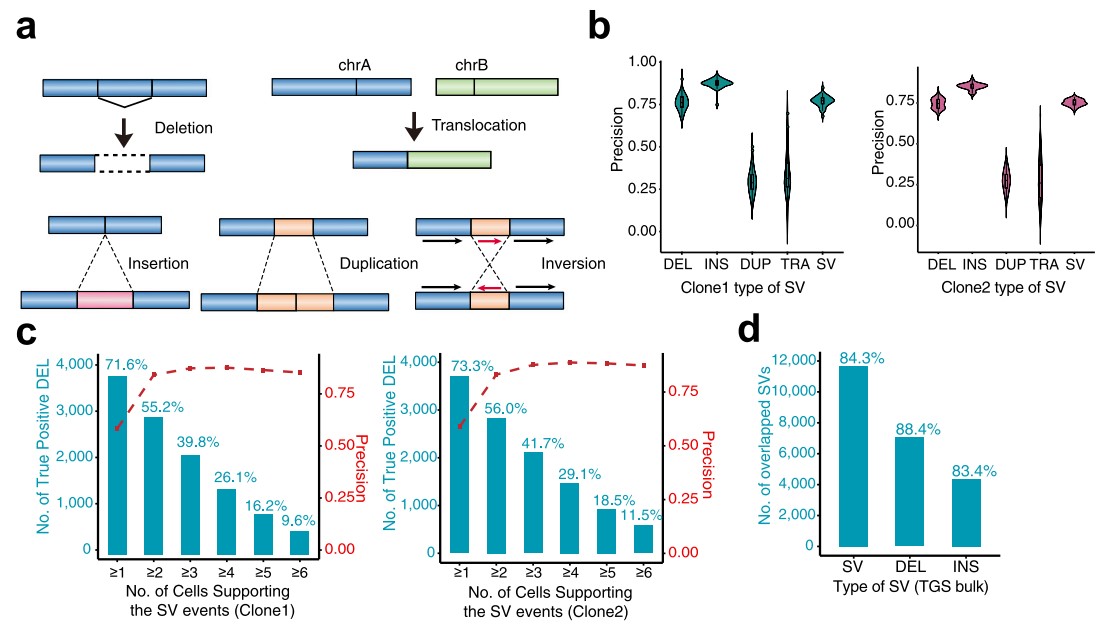
图2
3. 通过SMOOTH-seq检测K562中的ecDNA
EcDNA在致癌过程中非常常见,致癌基因可以在ecDNA中扩增。SMOOTH-seq的长读长可以在单次测序读取中捕获小于10kb的全长ecDNA。当只有一个拷贝的Tn5转座酶与ecDNA分子结合时,整个环状DNA分子可以被扩增成一个覆盖其全长的线性片段(图3a)。通过这种方式,可以检测到相同ecDNA的不同拷贝的完全相同长度的不同read,这是串联重复序列中不存在的特征(图3a)。作者从至少两个单独的K562细胞中确定了125个候选ecDNA(图3b)。这些ecDNA主要从5kb到1Mb(图3c),其中29.6%包含基因。90%候选ecDNA可以通过PCR结合Sanger测序进行验证(图3e)。对于8个小于15kb的候选ecDNA,作者使用不同的引物扩增了其全长序列(图3d)。8个候选ecDNA中有6个以成功扩增了预期的全长大小,通过PCR结合Sanger测序进一步验证环化位点(图3d、e)。综合来看,SMOOTH-seq可以准确检测单个细胞中的ecDNA。
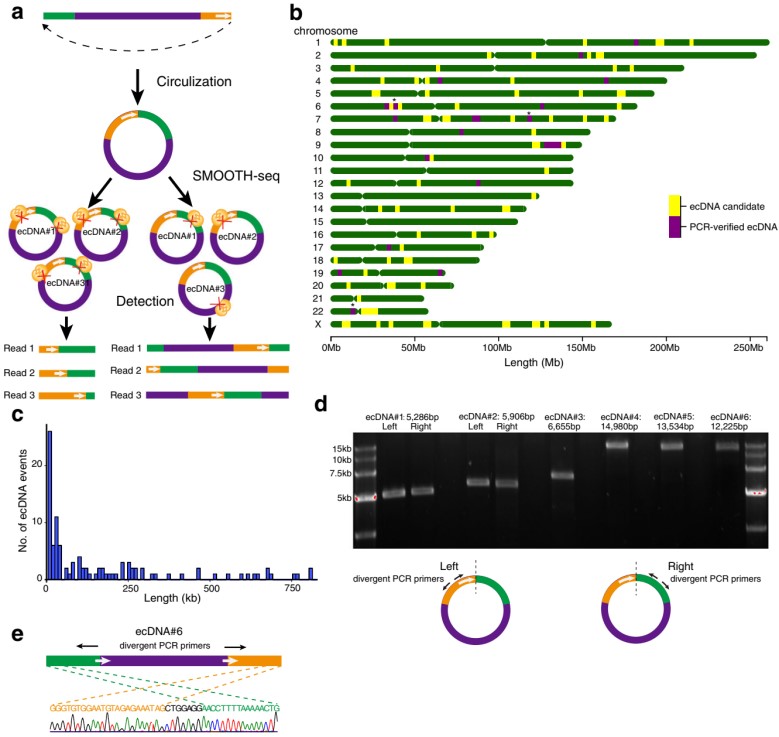
图3
4. 检测K562细胞中的SNV
在SNV检测方面,作者首先使用批量NGS测序数据作为对照,评估了在单个细胞中检出SNV的准确性。NGS样本中,总共检测了68,194和87,060个SNV,其中单个细胞平均覆盖率分别为7.5%和6.7%。每个单细胞SNV检测的假阳性率平均为2.0×10-5(图4a)。SMOOTH-seq表现出对CG-to-TA假阳性的偏好(图4b)。这种假阳性已在许多scWGS研究中有广泛报道,并且SMOOTH-seq比以前的scWGS方法有更好的表现。
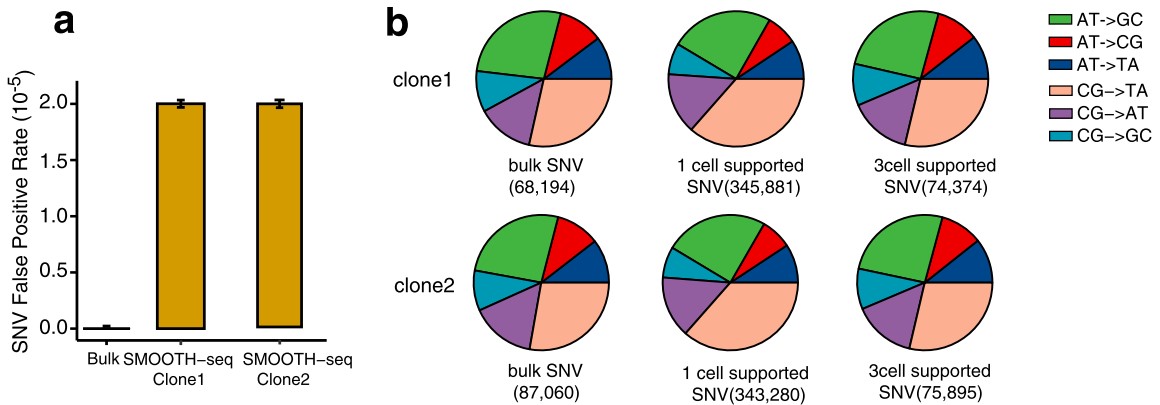
图4
5. 使用SMOOTH-seq在结肠癌细胞中检测SV
作者对一名结直肠癌患者的96个细胞进行了SMOOTH-seq,在单个细胞中检测到了SV(图5a)。去除与K562细胞中共有的突变,获得结直肠癌细胞特异性SV突变,共3570个SV事件(1376个插入、1661个缺失、230个易位和303个重复)(图5b),并能够识别超过5kb的重复事件(图5c-e)。研究发现在结直肠癌肿瘤样本和K562细胞系中共有的SV,可以在多个人类基因组DNA(肿瘤邻近的正常组织、B细胞系GM12878和来自另一个人的外周血单核细胞)样品中检测到。作者怀疑这是由于目前的人类基因组注释仍然不够完善,第三代测序将有助于组装出更完整的基因组参考序列。
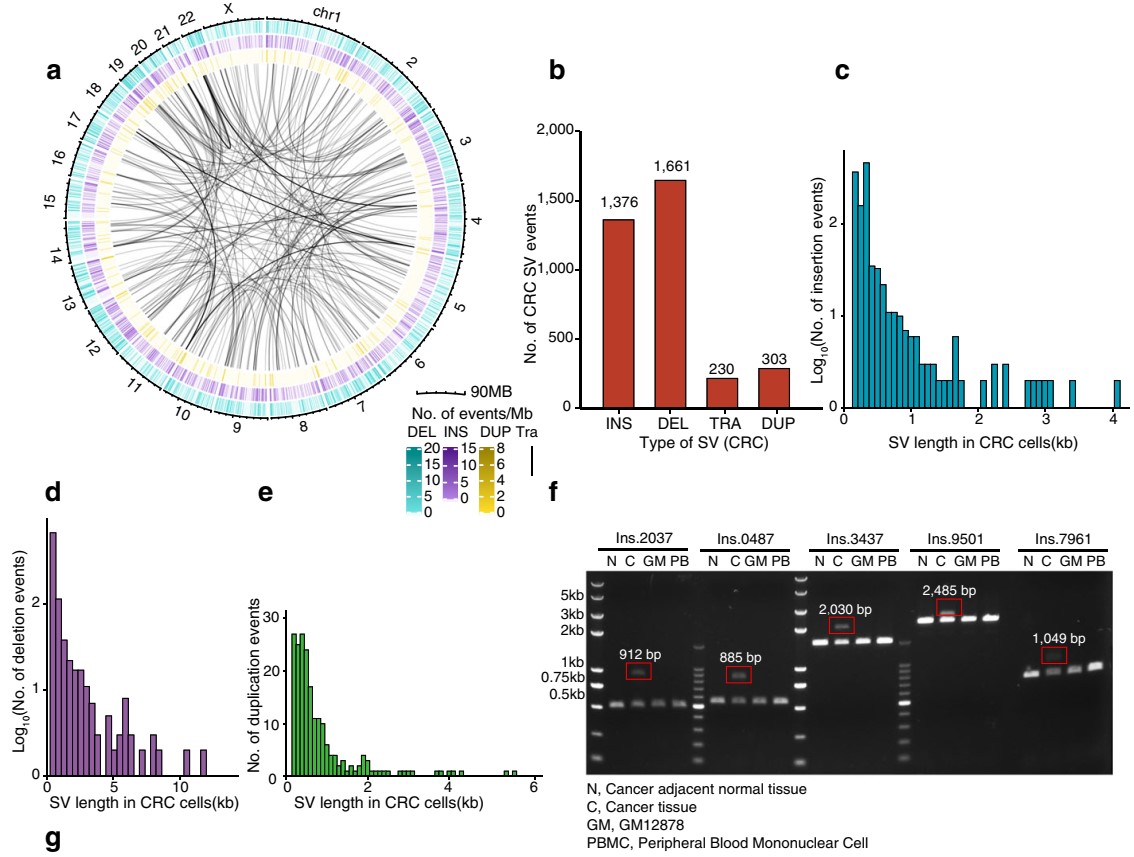
图5
总结
本文提出了一种基于第三代测序技术的单细胞基因组分析新方法SMOOTH-seq。利用读长长和高保真的优势,可以高精度检测基因组结构变异(特别是SV)、ecDNA等事件,该方法在单细胞基因组学领域具有广泛的应用前景。